摘要:理論物理學家對谷歌的人工智能AlphaFold進行了測試,發(fā)現(xiàn)了復雜的蛋白質(zhì)結(jié)。
加州大學戴維斯分校的研究人員已經(jīng)能夠在雞蛋中產(chǎn)生針對蛋白質(zhì)的化學成分,即氨基酸序列,如何決定其三維結(jié)構(gòu)的問題,是半個多世紀以來生物物理學面臨的大挑戰(zhàn)。這種關于所謂蛋白質(zhì)“折疊”的知識需求量很大,因為它對理解各種疾病及其治療等方面有重大貢獻。出于這些原因,谷歌的DeepMind研究團隊開發(fā)了AlphaFold,這是一款可以預測3D結(jié)構(gòu)的人工智能。
一個由美因茨約翰內(nèi)斯·古登堡大學(JGU)和加州大學洛杉磯分校的研究人員組成的團隊現(xiàn)在已經(jīng)仔細研究了這些結(jié)構(gòu),并研究了它們與繩結(jié)的關系。我們知道繩結(jié)主要來自鞋帶和電纜,但它們也會在我們細胞的納米尺度上發(fā)生。結(jié)結(jié)蛋白不僅可以用來評估結(jié)構(gòu)預測的質(zhì)量,還可以提出關于折疊機制和蛋白質(zhì)進化的重要問題。
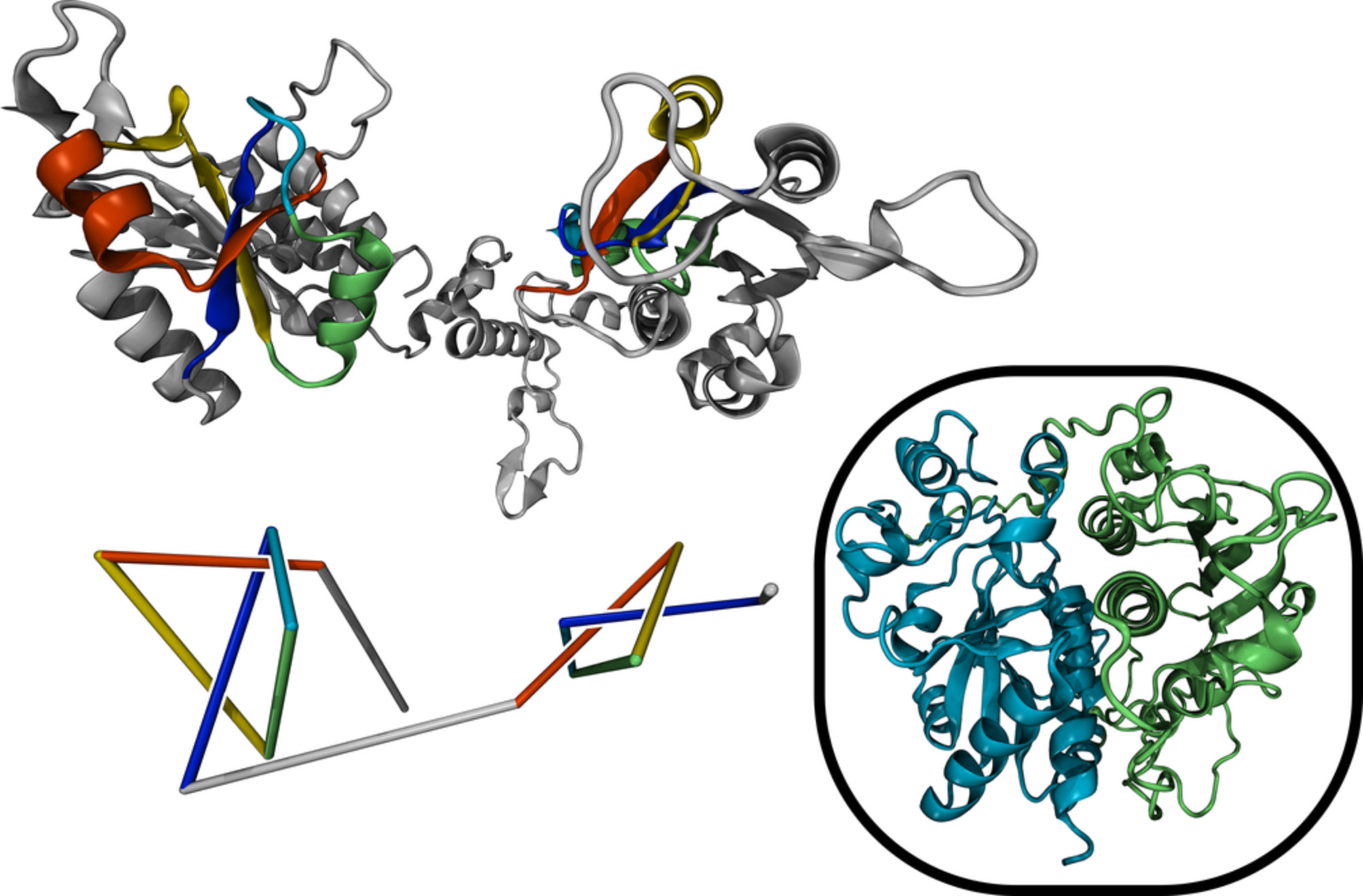
圖1 蛋白質(zhì)Q313J9(甲基轉(zhuǎn)移酶)中六交叉復合結(jié)的3D結(jié)構(gòu)(頂部)和簡化表示(底部)(來源:Wiley Online Library)
復雜的結(jié)作為AlphaFold的測試
美因茨大學(Mainz University)彼得·維爾諾(Peter Virnau)博士團隊的博士生Maarten a . Brems說:“我們對AlphaFold的所有新蛋白質(zhì)結(jié)預測進行了數(shù)值研究,大約有10萬個。”他們的目標是找出含有復雜且未知的蛋白質(zhì)結(jié)的稀有、高質(zhì)量結(jié)構(gòu),為AlphaFold的預測提供實驗驗證的基礎。這項研究不僅發(fā)現(xiàn)了復雜的結(jié)狀蛋白質(zhì),而且還發(fā)現(xiàn)了蛋白質(zhì)中的第一個復合結(jié)。后者可以被認為是同一根弦上的兩個不同的結(jié)。“這些新發(fā)現(xiàn)還為這種罕見蛋白質(zhì)背后的進化機制提供了深入的見解,”參與該項目的理論物理學家羅伯特·朗克爾(Robert Runkel)補充說。這項研究的結(jié)果發(fā)表在《蛋白質(zhì)科學》雜志上。
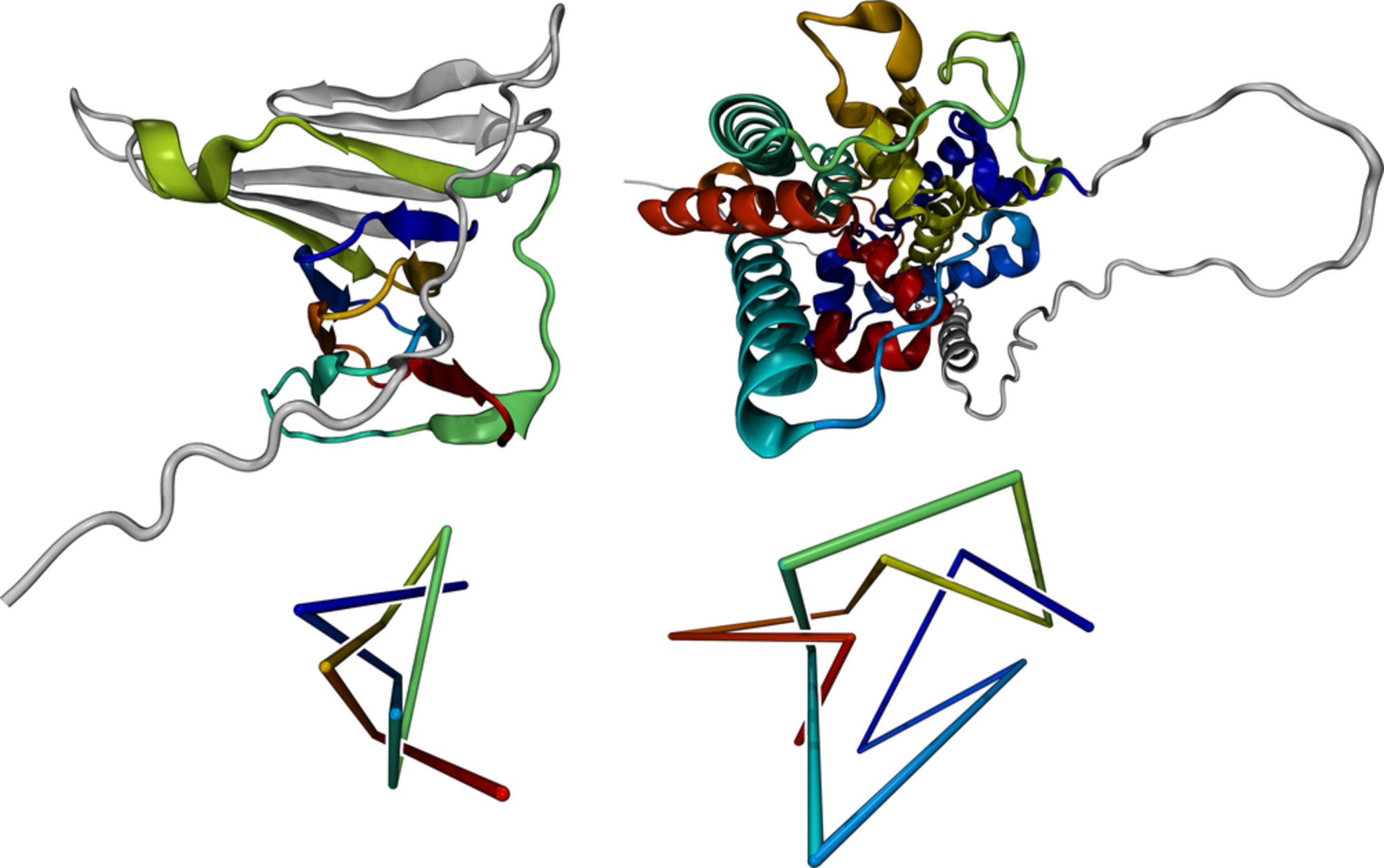
圖2 蛋白質(zhì)A0A0K0IQS9(左)和C1GYM9(右)的結(jié)構(gòu)和拓撲。上圖:AlphaFold預測的3D結(jié)構(gòu)。底部:簡化表示,分別顯示左側(cè)和右側(cè)結(jié)構(gòu)中的51節(jié)和52節(jié)(來源:Wiley Online Library)
Peter Virnau博士對結(jié)果很滿意:“我們已經(jīng)和來自加州大學洛杉磯分校的同事Todd Yeates建立了合作關系,通過實驗來確認這些結(jié)構(gòu)。這一研究方向?qū)⑺茉焐镂锢斫鐚θ斯ぶ悄艿目捶ā覀兒苄疫\能有像耶茨博士這樣的專家參與其中。”
參考資料:
1. Maarten A. Brems, Robert Runkel, Todd O. Yeates, Peter Virnau. AlphaFold predicts the most complex protein knot and composite protein knots. Protein Science, 2022; 31 (8) DOI: 10.1002/pro.4380
2. John Jumper, Richard Evans, Alexander Pritzel, Tim Green, Michael Figurnov, Olaf Ronneberger, Kathryn Tunyasuvunakool, Russ Bates, Augustin ?ídek, Anna Potapenko, Alex Bridgland, Clemens Meyer, Simon A. A. Kohl, Andrew J. Ballard, Andrew Cowie, Bernardino Romera-Paredes, Stanislav Nikolov, Rishub Jain, Jonas Adler, Trevor Back, Stig Petersen, David Reiman, Ellen Clancy, Michal Zielinski, Martin Steinegger, Michalina Pacholska, Tamas Berghammer, Sebastian Bodenstein, David Silver, Oriol Vinyals, Andrew W. Senior, Koray Kavukcuoglu, Pushmeet Kohli, Demis Hassabis. Highly accurate protein structure prediction with AlphaFold. Nature, 2021; 596 (7873): 583 DOI: 10.1038/s41586-021-03819-2
2. John Jumper, Richard Evans, Alexander Pritzel, Tim Green, Michael Figurnov, Olaf Ronneberger, Kathryn Tunyasuvunakool, Russ Bates, Augustin ?ídek, Anna Potapenko, Alex Bridgland, Clemens Meyer, Simon A. A. Kohl, Andrew J. Ballard, Andrew Cowie, Bernardino Romera-Paredes, Stanislav Nikolov, Rishub Jain, Jonas Adler, Trevor Back, Stig Petersen, David Reiman, Ellen Clancy, Michal Zielinski, Martin Steinegger, Michalina Pacholska, Tamas Berghammer, Sebastian Bodenstein, David Silver, Oriol Vinyals, Andrew W. Senior, Koray Kavukcuoglu, Pushmeet Kohli, Demis Hassabis. Highly accurate protein structure prediction with AlphaFold. Nature, 2021; 596 (7873): 583 DOI: 10.1038/s41586-021-03819-2
摘要:理論物理學家對谷歌的人工智能AlphaFold進行了測試,發(fā)現(xiàn)了復雜的蛋白質(zhì)結(jié)。
加州大學戴維斯分校的研究人員已經(jīng)能夠在雞蛋中產(chǎn)生針對蛋白質(zhì)的化學成分,即氨基酸序列,如何決定其三維結(jié)構(gòu)的問題,是半個多世紀以來生物物理學面臨的大挑戰(zhàn)。這種關于所謂蛋白質(zhì)“折疊”的知識需求量很大,因為它對理解各種疾病及其治療等方面有重大貢獻。出于這些原因,谷歌的DeepMind研究團隊開發(fā)了AlphaFold,這是一款可以預測3D結(jié)構(gòu)的人工智能。
一個由美因茨約翰內(nèi)斯·古登堡大學(JGU)和加州大學洛杉磯分校的研究人員組成的團隊現(xiàn)在已經(jīng)仔細研究了這些結(jié)構(gòu),并研究了它們與繩結(jié)的關系。我們知道繩結(jié)主要來自鞋帶和電纜,但它們也會在我們細胞的納米尺度上發(fā)生。結(jié)結(jié)蛋白不僅可以用來評估結(jié)構(gòu)預測的質(zhì)量,還可以提出關于折疊機制和蛋白質(zhì)進化的重要問題。

圖1 蛋白質(zhì)Q313J9(甲基轉(zhuǎn)移酶)中六交叉復合結(jié)的3D結(jié)構(gòu)(頂部)和簡化表示(底部)(來源:Wiley Online Library)
復雜的結(jié)作為AlphaFold的測試
美因茨大學(Mainz University)彼得·維爾諾(Peter Virnau)博士團隊的博士生Maarten a . Brems說:“我們對AlphaFold的所有新蛋白質(zhì)結(jié)預測進行了數(shù)值研究,大約有10萬個。”他們的目標是找出含有復雜且未知的蛋白質(zhì)結(jié)的稀有、高質(zhì)量結(jié)構(gòu),為AlphaFold的預測提供實驗驗證的基礎。這項研究不僅發(fā)現(xiàn)了復雜的結(jié)狀蛋白質(zhì),而且還發(fā)現(xiàn)了蛋白質(zhì)中的第一個復合結(jié)。后者可以被認為是同一根弦上的兩個不同的結(jié)。“這些新發(fā)現(xiàn)還為這種罕見蛋白質(zhì)背后的進化機制提供了深入的見解,”參與該項目的理論物理學家羅伯特·朗克爾(Robert Runkel)補充說。這項研究的結(jié)果發(fā)表在《蛋白質(zhì)科學》雜志上。

圖2 蛋白質(zhì)A0A0K0IQS9(左)和C1GYM9(右)的結(jié)構(gòu)和拓撲。上圖:AlphaFold預測的3D結(jié)構(gòu)。底部:簡化表示,分別顯示左側(cè)和右側(cè)結(jié)構(gòu)中的51節(jié)和52節(jié)(來源:Wiley Online Library)
Peter Virnau博士對結(jié)果很滿意:“我們已經(jīng)和來自加州大學洛杉磯分校的同事Todd Yeates建立了合作關系,通過實驗來確認這些結(jié)構(gòu)。這一研究方向?qū)⑺茉焐镂锢斫鐚θ斯ぶ悄艿目捶ā覀兒苄疫\能有像耶茨博士這樣的專家參與其中。”
參考資料:
1. Maarten A. Brems, Robert Runkel, Todd O. Yeates, Peter Virnau. AlphaFold predicts the most complex protein knot and composite protein knots. Protein Science, 2022; 31 (8) DOI: 10.1002/pro.4380
2. John Jumper, Richard Evans, Alexander Pritzel, Tim Green, Michael Figurnov, Olaf Ronneberger, Kathryn Tunyasuvunakool, Russ Bates, Augustin ?ídek, Anna Potapenko, Alex Bridgland, Clemens Meyer, Simon A. A. Kohl, Andrew J. Ballard, Andrew Cowie, Bernardino Romera-Paredes, Stanislav Nikolov, Rishub Jain, Jonas Adler, Trevor Back, Stig Petersen, David Reiman, Ellen Clancy, Michal Zielinski, Martin Steinegger, Michalina Pacholska, Tamas Berghammer, Sebastian Bodenstein, David Silver, Oriol Vinyals, Andrew W. Senior, Koray Kavukcuoglu, Pushmeet Kohli, Demis Hassabis. Highly accurate protein structure prediction with AlphaFold. Nature, 2021; 596 (7873): 583 DOI: 10.1038/s41586-021-03819-2
2. John Jumper, Richard Evans, Alexander Pritzel, Tim Green, Michael Figurnov, Olaf Ronneberger, Kathryn Tunyasuvunakool, Russ Bates, Augustin ?ídek, Anna Potapenko, Alex Bridgland, Clemens Meyer, Simon A. A. Kohl, Andrew J. Ballard, Andrew Cowie, Bernardino Romera-Paredes, Stanislav Nikolov, Rishub Jain, Jonas Adler, Trevor Back, Stig Petersen, David Reiman, Ellen Clancy, Michal Zielinski, Martin Steinegger, Michalina Pacholska, Tamas Berghammer, Sebastian Bodenstein, David Silver, Oriol Vinyals, Andrew W. Senior, Koray Kavukcuoglu, Pushmeet Kohli, Demis Hassabis. Highly accurate protein structure prediction with AlphaFold. Nature, 2021; 596 (7873): 583 DOI: 10.1038/s41586-021-03819-2



